摘 要: 分子育种是世界农作物育种的发展方向。综述了利用第 3 代新型分子标记研制的 SNP 芯片所具有的高集成、高通量、微型化和自动化等优点,介绍了基于 Illumina 公司的 BeadArray 技术和 Affymetrix 公司的 Axiom 技术开发的相应芯片鉴定平台及其在水稻、小麦、玉米等粮食作物和棉花、花生、向日葵等经济作物育种过程中的应用情况,讨论了 SNP 芯片技术在应用中存在的问题,并对其前景进行了展望。
关键词: SNP;芯片技术;农作物;分子育种
全球人口将在 2050 年突破 90 亿,而目前主要农作物的产量已经无法满足人口的快速增长带来的对粮食的巨大需求 [1] ,加上世界耕地面积不断减少、全球变暖可能引发的气候异常或病虫害暴发等因素,农作物育种在未来几十年内面临着严峻挑战 [2] .因此,充分利用目前作物基因组学取得的研究成果,将先进的分子生物技术与常规育种技术相结合,从本质上推动作物育种领域的绿色革命,对缓解甚至解决全球粮食安全问题具有重要意义。
1 育种分子标记
作物分子育种的核心是基于分子标记的辅助选择(marker- assisted selection,MAS),分子标记最早始于以限制性片段长度多态性(Restriction Frag-ment Length Polymorphism,RFLP) 为代表的第 1 代分子标记,随后发展出目前应用最广的第 2 代标记-- - 简单重复序列 (Simple Sequence Repeats,SSR)标记,在育种领域发挥了巨大作用,但随着育种精度要求的不断提高,以及小麦等多倍体作物分子育种工作的开展,SSR 标记开始出现局限性。一是 SSR 序列在基因组中的分布不均匀且是有限的,其与目标基因连锁不紧密,容易在分子辅助选择过程中丢失;二是 SSR 标记依赖于凝胶电泳技术,用于规模育种中则通量低、代价高;三是 SSR 标记在多倍体中的亚基因组特异性较差,会影响图谱构建或基因定位结果的准确性。
随着测序和组装技术的进步,越来越多的农作物物种已经绘制全基因组草图。基于基因组数据,科研人员诊断、开发了第 3 代新型标记-- - 单核苷酸多态性(Single Nucleotide Polymorphism,SNP)标记,SNP 标记具有密度高、遗传稳定的特点,而且几乎都以二等位多态性存在 [3] ,检测时无须象 RFLP,SSR 那样对片段的长度作出测量,极易实现自动化。
2 芯片技术平台
SNP 芯片技术就是将第 3 代 SNP 标记固定在载体上形成密集的寡核苷酸探针阵列,再与目标DNA 进行等位基因特异性反应,根据反应后信号的有无和强弱确定 SNP 位点的多态性,该技术可以对作物全基因组实现快速、高密度的扫描,特别是对育种工作中的大量群体样本进行基因型鉴定时,单个检测位点成本很低,是一种高集成、高通量、微型化和自动化的检测 SNP 的手段。目前,芯片基因型鉴定平台采用的是 Illumina 公司的 BeadArray 技术和 Affymetrix公司的 Axiom技术。
Illumina BeadArray 技术是将特定寡核苷酸覆盖到微珠上,将其嵌入到装有样本 DNA 的、能与其互相匹配的微孔中发生杂交,再通过荧光扫描实现对 SNP 的高通量识别和分型。BeadArray技术可以分析更高密度的阵列,即能分析更多的 SNP 位点。基于双色单碱基探针,每个样本可以同时扫描 3 000到 500 多万个 SNP 位点。Affymetrix则采用了基于光蚀刻的 GeneChip 阵列与等位基因特异性寡聚体进行杂交,该寡聚体包含了能与 SNP 完全匹配的探针 [4] .AffymetrixAxiom技术基于双色和 30- mer 探针的连接测定,可同时分析 384 个样品的 50K 个 SNP位点。
3 芯片技术在育种中的应用
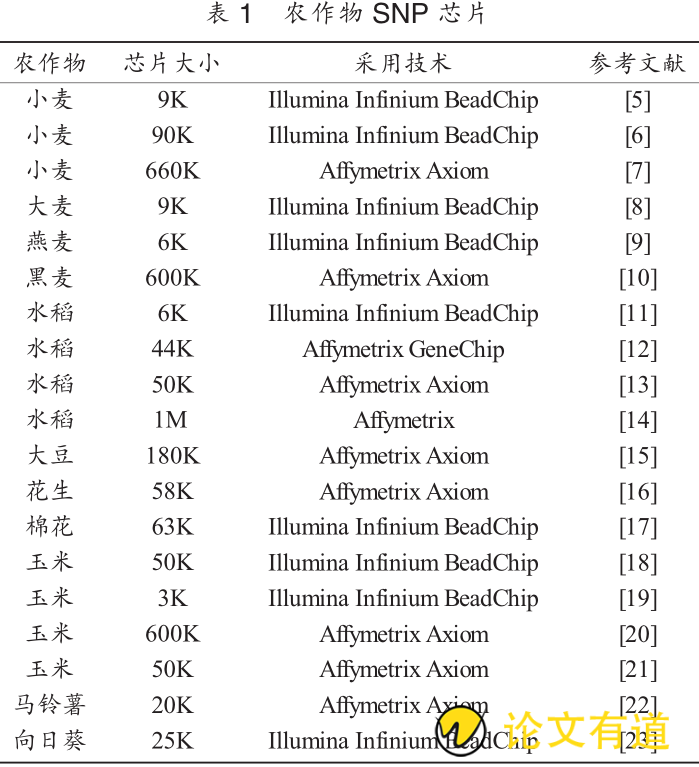
水稻是最早完成全基因组测序的农作物,因此,其芯片研发工作也起步最早。目前,已经开发出6K,44K,50K和 1M等满足不同育种用途的 SNP 芯片(表 1)。在其他粮食作物中,玉米 3K/50K/600K、大豆 180K、马铃薯 20K芯片也先后研发成功。小麦9K/90K/660K、大麦 9K、燕麦 6K 和黑麦 600K 等麦类作物芯片也已用于生产。而在经济作物中,目前已研制出花生 58K、棉花 63K 和向日葵 25K 芯片用于育种(表 1)。
芯片技术已被广泛用于育种领域中的各个环节中,并且根据各环节实际需求开发出了不同大小的芯片以及鉴定平台。例如,在全基因组关联分析(Genome- wide Association Studies,GWAS)和连锁作图中需要对数千个样本扫描数万个标记,而在标记辅助回交(Marker- assisted Backcrossing,MABC)中只需要对样本扫描数百个与目标基因紧密连锁的标记。因此,在作物分子育种中,一般先利用高密度芯片快速准确地定位目标基因,对于多基因控制的农艺性状则确定其数量性状位点,而在接下来的回交育种和轮回选择等工作中,可使用更具有针对性的、基于竞争性等位基因特异性 PCR(KompetitiveAllele Specific PCR,KASP)的 SNP 芯片对后代单株进行选择。此外,研究人员还开发出了基于芯片技术的分子育种工具 MBDT(Marker- assisted Back-cross BreedingTool)。该工具包括数据验证、表型分析、连锁图谱构建、QTL 分析、基因组显示和 MABC所需样本量,使目前先进的分子育种技术操作简单化、结果直观化,将大大提高传统育种家使用 SNP芯片的比例,推动农业作物进入以科学理论为基础的分子育种时代。
4 存在问题和展望
在水稻、玉米等二倍体模式作物中,对基因组以及基因功能的研究较为深入,分子育种也取得了很好的成果。而在燕麦等冷门作物或者小麦等具有复杂基因组的多倍体作物中,基于全基因组水平的功能基因挖掘与验证工作才刚刚起步,在利用芯片技术选育品种过程中很可能会出现 2 个问题。
第 1 个问题是作物基因型与预期表型不一致,这也是限制分子育种应用的一个重要因素。阐明控制作物产量、品质、抗性等重要农艺性状的基因及其调控机制,是分子育种的理论基础,对此,科研人员开展多方面的研究,如泛基因组重测序、与模式作物比较基因组学分析、育种环节结合目标基因功能验证、引入转录组学 / 蛋白组学 / 代谢组学的研究成果等等,以期减少或消除基因型与表型不一致的情况。
第 2 个问题是芯片标记信息不全面。由于芯片标记主要是基于已测序作物品种的基因组序列开发的,而在育种过程中常常会引入作物的农家品种、野生品种甚至近缘种的优异基因组片段,因此,现有育种芯片可能无法识别作物不同遗传资源中的特有变异类型,最终限制了外源片段的导入。例如,早期基于 B73 和 Mo17 这 2 个温带玉米自交系测序草图开发的 SNP 芯片,无法识别出热带玉米基因组中的优异等位变异 [24] ,为此,已有机构开始对包括热带自交系在内的玉米品种进行大规模重新测序,以期开发出信息量完整的 SNP 芯片用于育种。
在接下来的 10 a 内,随着组学技术的进一步发展和功能研究的不断深入,SNP 芯片的成本将继续降低,功能标记的准确性和覆盖品种的完整性则会大幅提升,鉴定平台和育种软件的简洁化、智能化将使芯片技术在传统育种家以及发展中国家大范围普及,世界农业将真正进入科学、高效的分子育种时代。
参考文献:
[1] RAY D K,RAMANKUTTY N,MUELLER N D,et al. Recent pat-terns ofcrop yield growth and stagnation [J]. Nat Commun,2012,3:1293.
[2]ABBERTON M,BATLEY J,BENTLEY A,et al. Global agriculturalintensification during climate change: a role for genomics [J]. PlantBiotechnol J,2015,14(4):1095- 1098.
[3] BROOKES A J. The essence of SNPs [J]. Gene,1999,234(2):177- 186.
[4] MATSUZAKI H,DONG S,LOI H,et al. Genotyping over 100,000SNPs on a pair of oligonucleotide arrays [J]. Nat Methods,2004,1(2):109- 111.
[5] CAVANAGH C R,CHAO S M,WANG S C,et al. Genome- widecomparative diversity uncovers multiple targets of selection for im-provement in hexaploid wheat landraces and cultivars [J]. Proc NatlAcad Sci USA,2013,110(20):8057- 8062.
[6]WANG Y,CHENG X,SHAN Q,et al. Simultaneous editing of threehomoeoalleles in hexaploid bread wheat confers heritable resistancetopowderymildew[J]. Nat Biotechnol,2014,32(9):947- 951.
[7]赵光耀,孔秀英,高丽峰,等。 小麦基因组特异 SNP 分析与高通量芯片开发 [C]// 第五届全国小麦基因组学及分子育种大会会议论文集,合肥:中国作物协会,2014.
[8]COMADRAN J,KILIAN B,RUSSELL J,et al. Natural variation in ahomolog of Antirrhinum CENTRORADIALIS contributed to springgrowth habit and environmental adaptation in cultivated barley[J].Nat Genet,2012,44(12):1388- 1392.
[9]TINKER N A,CHAO S,LAZO G R,et al. A SNP genotyping arrayfor hexaploid oat[J]. Plant Genome,2014,7:3.
[10] BAUER E,SCHMUTZER T,BARILAR I,et al. Towards awhole- genome sequence for rye(Secale cereale L. )[J]. Plant J,2017,89(5):853- 869.
[11]YU H,XIE W,LI J,et al. A whole- genome SNP array(RICE6K)for genomic breeding in rice [J]. Plant Biotech J,2014,12(1):28- 37.
[12] TUNG C W,ZHAO K,WRIGHT M,et al. Development of a re-search platform for dissecting phenotype- genotype associations inrice (Oryza spp. )[J]. Rice,2010,3(4):205- 217.
[13] SINGH N,JAYASWAL P K,PANDA K,et al. Single- copy genebased 50K SNP chip for genetic studies and molecular breeding inrice[J]. Sci Rep,2015,5:11600.
[14] MCCOUCH S R,ZHAO K,WRIGHT M,et al. Development ofgenome- wide SNP assays for rice [J]. Breed Sci,2010,60:524- 535.
[15]LEE Y G,JEONG N,KIM J H,et al. Development,validation andgenetic analysis of a large soybean SNP genotyping array [J]. PlantJ,2015,81(4):625- 636.
[16]PANDEY MK,AGARWAL G,KALE S M,et al. Development andevaluation of a high density genotyping' Axiom_Arachis' array with58K SNPs for accelerating genetics and breeding in groundnut[J].Sci Rep,2017,7:40577.
[17]HULSE- KEMP AM,LEMMJ,PLIESKE J,et al. Development ofa63KSNP arrayfor cotton and high- densitymappingofintraspecificand interspecific populations ofGossypium spp.[J]. G3(Bethesda),2015,5(6):1187- 1209.
[18]GANAL MW,DURSTEWITZ G,POLLEY A,et al. A large maize(Zea mays L. )SNP genotyping array:development and germplasmgenotyping,and genetic mappingtocompare with the B73 referencegenome[J]. PLoSOne,2011,6(12):e28334.
[19]ROUSSELLE Y,JONES E,CHARCOSSET A,et al. Study on es-sential derivation in maize:III. Selection and evaluation of a panelof single nucleotide polymorphism loci for use in European andNorth American germplasm [J]. Crop Sci,2015,55 (3):1170- 1180.
[20] UNTERSEER S,BAUER E,HABERER G,et al. A powerful toolfor genome analysis in maize: development and evaluation of thehigh density 600k SNP genotyping array [J]. BMC Genomics,2014,15(1):823.
[21]XUC,RENY,JIANY,et al. Development ofa maize 55KSNP ar-raywith improved genome coverage for molecular breeding [J]. MolBreed,2017,37(31):20.
[22]VOSP G,UITDEWILLIGENJ G,VOORRIPSR E,et al. Develop-ment and analysis of a 20K SNP arrayfor potato(Solanum tubero-sum):an insight into the breeding history [J]. Theor Appl Genet,2015,128(12):2387- 2401.
[23] LIVAJA M,UNTERSEER S,ERATH W,et al. Diversity analysisand genomic prediction of Sclerotinia resistance in sunflower usinga new25KSNP genotypingarray [J]. Theor Appl Genet,2016,129(2):317- 329.
[24]XU Y,LI P,ZOU C,et al. Enhancing genetic gain in the molecularbreedingera[J]. J Exp Bot,2017,68(11):2641- 2666.

